Génomes mitochondriaux des mammifères
En 1963, l’ADN a d’abord été détecté dans les mitochondries (N.M. K. Nass et S. Nass, 1963). Au cours des 30 années suivantes, la séquence complète de l’ADN mitochondrial (ADNmt) [environ 17 000 paires de bases (pb)] a été déterminée chez plus d’une douzaine d’espèces, y compris les humains (Anderson et al., 1981).
La plupart des cellules de vertébrés en culture semblent avoir environ 1000-5000 molécules du génome mitochondrial circulaire (Bogenhagen et Clayton, 1974, Shmookler Reis et Goldstein, 1983). L’ADNmt se localise dans la matrice mitochondriale et semble être associé aux protéines et aux lipides (Hillar et al., 1979). Chez la levure, les plus grands génomes mitochondriaux de ~ 80 kilobases (kb) sont organisés en 10-20 nucléoïdes distincts (c’est-à-dire des complexes protéine-ADN), sphériques ou ovoïdes, mesurant 0,3-0,6 μ de diamètre.
Les nucléoïdes contiennent entre 3 et 4 génomes mitochondriaux et jusqu’à 20 polypeptides différents (Miyakawa et al., 1987, Kaufman et al., 2000).
On ne sait pas si l’ADNmt des animaux est également organisé sous la forme de complexes ADN-protéine (nucléoïdes), bien que cette possibilité ait été suggérée comme un système pour maintenir la stabilité génétique (Jacobset al., 2000). Une des différences les plus frappantes entre la levure et les systèmes animaux peut être observée au cours du développement des cellules animales. Le nombre de copies d’ADN mitochondrial semble être strictement contrôlé pendant le développement (Piko et Taylor, 1987, Lefai et al., 2000b), et un mécanisme spécifique peut avoir évolué en raison de ces besoins. L’identification récente d’une hélicase mitochondriale appelée Twinkle, qui montre une localisation ponctuée compatible avec une structure nucléoïde, vient appuyer ce modèle (Spelbrink et al., 2001).
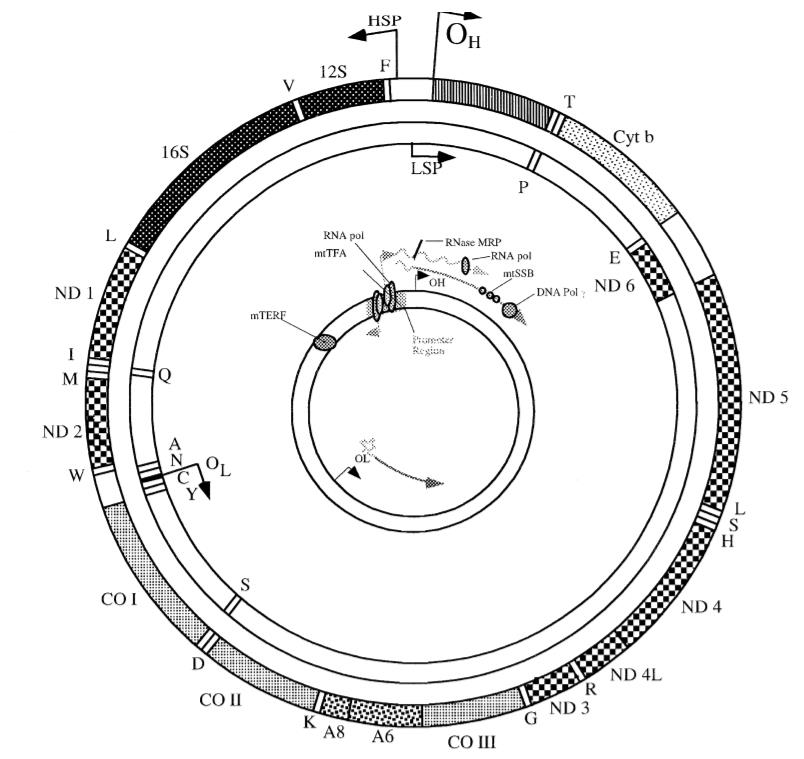
Structure de l’ADN mitochondrial humain. Cette figure représente l’ADNmt humain de 16 569 pb montrant 13 gènes codant pour une protéine ainsi que 2 gènes codant pour l’ARNr et 22 ARNt. Les gènes codant pour les sous-unités du complexe I (ND1-ND6), du complexe III (Cyt b), du complexe IV (COX I-COX III) et du complexe V (A8 et A6) sont montrés par différentes trappes. L’insert dans cette figure illustre les mécanismes associés à la réplication et à la transcription de l’ADNmt, y compris les sites de liaison pour l’ARN polymérase mitochondriale, le facteur de transcription mitochondrial mtTFA, l’enzyme de traitement de l’ARN RNAse MRP et le facteur de terminaison de la transcription mTERF. Les origines de la réplication pour les brins H- et L- (OH et OL) sont également montrées.
Les analyses par microscopie électronique ont montré que l’ADNmt des mammifères peut être arrangé en tant que monomères unicirculaires, mais aussi en tant que dimères unicirculaires ou formes caténatées (Clayton, 1982). Ces premières études ont également montré ce que l’on a appelé la boucle de déplacement ou « boucle D » comme une séparation des brins dans une région spécifique de l’ADNmt. On sait maintenant que la plupart des séquences associées à l’initiation de la replication ou de la transcription de l’ADNmt sont à proximité de la région de la boucle D (Clayton, 1982). La transcription et la replication d’un brin et la transcription du brin complémentaire sont toutes deux déclenchées à proximité de la boucle D. Ce segment d’ADN de 1123 pb est souvent dans une configuration à simple brin et contient des sites pour les protéines se liant à l’ADN qui contrôlent la réplication et la transcription de l’ADNmt. Il a été observé que des mutations dans cette région s’accumulent au cours du vieillissement (Michikawa et al., 1999, Wang et al., 2001), mais on ne sait pas encore si ces altérations affectent la réplication de l’ADNmt ou l’expression génique.
L’ADNmt humain
L’ADNmt humain est représentatif des génomes mitochondriaux des mammifères. Il s’agit d’une molécule circulaire double brin de 16 569 pb codant pour 13 polypeptides (figure ci-dessus). Tous les polypeptides codés par l’ADNmt sont des membres des complexes de phosphorylation oxydative (OXPHOS). Ils comprennent sept sous-unités du complexe I, une sous-unité du complexe III, trois sous-unités du complexe IV et deux du complexe V. Outre les gènes codant pour la protéine, l’ADNmt code également 22 ARNs de transfert (ARNt) et deux ARN ribosomaux (ARNs 12S et 16S ). L’expression et le maintien de l’ADN mitochondrial dépendent d’un grand nombre de facteurs nucléaires codés dans les ribosomes cytosoliques comme polypeptides précurseurs et importés dans les mitochondries via des pores d’importation spécialisés (Attardi et Schatz, 1988). Bien que les sous-unités catalytiques du système OXPHOS soient codées par l’ADNmt, ces complexes enzymatiques contiennent également un grand nombre de sous-unités codées par le noyau qui sont nécessaires à leur fonction.
La distribution asymétrique de la guanine et de la cytosine permet la séparation de l’ADNmt en brins « lourds » (brin H) et « légers » (brin L) dans la centrifugation en gradient de densité alcaline. Les ARNr, tous sauf un polypeptide, et 14 des 22 ARNt sont codés dans les gènes des brins lourds. Contrairement à l’ADNmt de Saccharomyces cerevisiae, l’ADNmt des vertébrés est dépourvu d’introns (Anderson et al., 1981). Il y a très peu de régions intergéniques non codantes, à l’exception de la région régulatrice contenant les promoteurs et l’origine de la réplication des brins lourds. L’information génétique est si condensée qu’il y a un chevauchement dans certaines séquences codantes, et des codons de terminaison peuvent être générés par l’addition d’adénines au transcrit pendant la polyadénylation des ARNm (Anderson et al., 1981). Le code génétique de l’ADN mitochondrial des vertébrés diffère du code nucléaire-cytoplasmique. Au lieu d’être un codon de terminaison, TGA code pour le tryptophane dans les mitochondries des vertébrés. L’ATA code pour la méthionine dans les mitochondries, mais l’isoleucine dans le cytosol. Enfin, AGA ou AGG dans les mitochondries codent pour un codon stop au lieu de l’arginine (Tableau ci-dessous) (Anderson et al., 1981).
Code génétique mitochondrien chez les mamfères
les acides aminés entre parenthèses correspondent au code génétique universel
| UUU | Phe | UCU | Ser | UAU | Tyr | UGU | Cys | |||
| UUC | Phe | UCC | Ser | UAC | Tyr | UGC | Cys | |||
| UUA | Leu | UCA | Ser | UAA | Ter | UGA | Trp(Ter) | |||
| UUG | Leu | UCG | Ser | UAG | Ter | UGG | Trp | |||
| CUU | Leu | CCU | Pro | CAU | His | CGU | Arg | |||
| CUC | Leu | CCC | Pro | CAC | His | CGC | Arg | |||
| CUA | Leu | CCA | Pro | CAA | Gln | CGA | Arg | |||
| CUG | Leu | CCG | Pro | CAG | Gln | CGG | Arg | |||
| AUU | Ile | ACU | Thr | AAU | Asn | AGU | Ser | |||
| AUC | Ile | ACC | Thr | AAC | Asn | AGC | Ser | |||
| AUA | Met(Ile) | ACA | Thr | AAA | Lys | AGA | Ter(Arg) | |||
| AUG | Met | ACG | Thr | AAG | Lys | AGG | Ter(Arg) | |||
| GUU | Val | GCU | Ala | GAU | Asp | GGU | Gly | |||
| GUC | Val | GCC | Ala | GAC | Asp | GGC | Gly | |||
| GUA | Val | GCA | Ala | GAA | Glu | GGA | Gly | |||
| GUG | Val | GCG | Ala | GAG | Glu | GGG | Gly |
Structure de la région D-Loop de l’ADNmt humain
La comparaison des séquences nucléotidiques de l’ADN mitochondrial des mammifères a révélé un certain degré de conservation dans les régions promotrices ainsi que dans trois autres régions (appelées Conserved Sequence Blocks ou CSB I, CSB II et CSB III) (Walberg et Clayton, 1981). Ces séquences sont conservées dans les régions en boucle D de nombreux vertébrés, suggérant des rôles importants pour ces motifs. On suppose que les CSB sont impliqués dans certains aspects de la replication de l’ADNmt parce qu’ils sont situés dans la région de la boucle D, et dans le cas de la CSB I, presque toujours près du site d’initiation de la synthèse de l’ADN.
Cependant, l’absence de certains CSB chez certains vertébrés suggère que la fonction de ces éléments peut être évitée par des configurations de régions de boucle D spécifiques ou que d’autres séquences nucléotidiques (ou facteurs protéiques) peuvent fournir la même fonction dans ces organismes. La majorité de la région de la boucle D contient des séquences non codantes et inclut des régions hypervariables (Greenberg et al., 1983). Bien que le taux global de mutations dans ces régions hypervariables soient nettement plus élevés que dans le reste de l’ADNmt (Greenberg et al., 1983), des positions de nucléotides semblent être des points chauds pour les changements (Stoneking, 2000). Les deux segments hypervariables (HV1 et HV2, positions 16024-16383 et 57-372, respectivement) ont été très utiles dans l’étude de l’évolution des eucaryotes (Lang et al., 1999), et plus spécifiquement, des populations humaines (Jorde et al. , 2000).
Réplication de l’ADN mitochondrial
Dans la plupart des cas, la réplication de l’ADN mitochondrial chez les mammifères est un processus asynchrone, commençant à l’origine de la réplication du brin H (OH) et se déroulant aux deux tiers du génome mitochondrial, jusqu’à l’origine de la réplication du brin L (Fig. 1) est forcé dans une configuration à simple brin par le brin H en extension. À ce stade, le brin H déplacé commence à être copié dans le brin L de la fille. La cartographie précise des espèces d’ARN et d’ADN dans la région de la boucle D a prouvé que l’ARN dérivé du promoteur L-strand (LSP) sert d’amorce pour la réplication de l’ADN du brin H (Chang et Clayton, 1985; 1985). Il existe également des preuves suggérant que les CSB sont impliqués dans la formation d’une amorce d’ARN correctement configurée. Cet ARN synthétisé à partir du promoteur du brin L (ARN 7S) est correctement traité par une activité de traitement de l’ARN mitochondrial (MRP, voir ci-dessous).
L’existence d’un hybride ARN-ADN en aval du LSP humain a été démontrée (Xu et Clayton, 1996). L’hybride humain est confiné à une région spécifique de l’origine (couvrant CSB II et CSB I), et la formation hybride est pratiquement abolie par des mutations dans l’élément CSB III en amont.
Une étude détaillée de l’hybride ARN-ADN de mammifères utilisant OH de souris a révélé une structure inhabituelle, où une molécule contenant une boucle R extrêmement stable constituée de deux brins d’ADN et un brin d’ARN (avec un des brins d’ADN déplacé par la molécule d’ARN hybride ) (Lee et Clayton, 1996). Comme cela a également été observé en utilisant une construction de plasmide contenant la région OH isolée, la séquence d’acide nucléique de OH de souris semble contenir toutes les informations requises pour la formation d’un hybride ARN-ADN stable.
Pour fournir une amorce appropriée pour la réplication, une endoribonucléase de traitement de l’ARN mitochondrial spécifique au site (RNase MRP) traite les substrats d’ARN 7S aux sites qui correspondent à certains des sites de transition ARN-ADN (c.-à-d. l’origine du brin H in vivo (Dairaghi et Clayton, 1993). L’enzyme MRP contient, en plus des composants protéiques, un ARN essentiel à l’activité (Chang et Clayton, 1987, Chang et Clayton, 1989). Le schéma de clivage de l’ARN par RNase MRP est cohérent avec le rôle de l’enzyme dans la fourniture d’amorces pour la réplication de l’ADNmt, le substrat étant probablement l’hybride ARN-ADN triple brin, plutôt que l’ARN simple brin. Ces clivages semblent dépendre de la présence de CSB I, ce qui suggère que la RNase MRP est nécessaire pour le traitement qui produit les amorces d’ARN dans les mitochondries de mammifères. Ces découvertes impliquent également l’hybride ARN-ADN comme substrat pour le traitement de l’ARN qui conduit à la formation des amorces pour la réplication du brin H.
L’activité MRase RNase est également trouvée dans le noyau de la cellule (Chang et Clayton, 1987, Gold et al., 1989). Plusieurs éléments de preuve ont démontré que la MRase nucléaire MRP est impliquée dans les étapes tardives du traitement de l’ARNr 5.8S dans le noyau (Lygerou et al., 1996). Cependant, une petite quantité d’ARN MRP a été localisée sur la mitochondrie dans des cellules de souris (Li et al., 1994) et des oocytes de Xenopus laevis (Davis et al., 1995) par hybridation in situ. De plus, des mutations dans les gènes codant pour le composant ARN de Saccharomyces cerevisiae et de l’ARN MRP de S. pombe RNase ont été isolées, ce qui aboutit à un phénotype mitochondrial (Paluh et Clayton, 1996). Prises ensemble, ces observations suggèrent l’existence d’un plus grand pool de RNAse MRP dans le noyau de la cellule qui est responsable du traitement de l’ARNr, et un pool plus petit dans les mitochondries qui semble être impliqué dans le traitement des amorces ARN mitochondriales.
Initiation de la réplication de l’ADN du brin L
Chez les vertébrés, l’origine de la réplication du brin L (OL) est située à une grande distance de OH sur la molécule d’ADNmt (Figure 1). L’initiation de la réplication de l’ADN du brin L a été étudiée chez les mammifères, où elle se produit dans une petite région non codante de [30 paires de bases (pb)] qui est flanquée de gènes d’ARNt. La séquence d’ADN dans cette région a le potentiel de supposer une structure tige-boucle stable (Tapper et Clayton, 1981) qui se formerait après que la fourche de réplication de l’initiation de la synthèse du brin H passe OL et expose le brin H parental dans cette région en tant que simple brin. La plupart de notre compréhension de la fonction des OG provient d’études qui ont utilisé un système de réplication in vitro pour l’OG humaine. Ces études ont montré que l’OL est capable d’initier la synthèse de l’ADN du brin L sur des sites correspondant à ceux cartographiés à partir d’acides nucléiques isolés des mitochondries in vivo (Wong et Clayton, 1985). L’initiation de la synthèse de l’ADN du brin L nécessite une ADN primase responsable de la génération de courtes molécules d’ARN avec 5 extrémités qui correspondent à la partie riche en T de la boucle dans la structure tige-boucle OL prédite. Chez la plupart des vertébrés, une région non codante avec une structure secondaire prédite conservée se trouve dans la séquence de la molécule d’ADNmt qui code un groupe d’ARNt pour les acides aminés Trp, Ala, Asn, Cys et Tyr (Anderson et al., 1981).
Caractéristiques générales des facteurs associés à la réplication mtDNA
De nombreux facteurs impliqués dans la réplication de l’ADN mitochondrial des mammifères ont été caractérisés.
Ceux-ci comprennent les sous-unités A (catalytique, également appelée sous-unité α) et B (accessoire, également appelée sous-unité β) de l’ADN polymérase γ, l’ARN polymérase mitochondriale, la protéine de liaison mitochondriale monobrin (mtSSB), le facteur de transcription mitochondriale A (mtTFA) et les enzymes de traitement de l’ARN (voir Lecrenier et Foury, 2000, Shadel et Clayton, 1997). Une ADN ligase mitochondriale, apparemment apparentée à l’ADN ligase III nucléaire, susceptible de participer à la résolution de brins répliqués a également été caractérisée (Pinz et Bogenhagen, 1998, Lakshmipathy et Campbell, 1999). Même si les fonctions primaires de ces facteurs sont comprises, leur rôle dans la régulation du nombre de copies d’ADNmt ne l’est pas.
De plus, les hélicases mitochondriales (Spelbrink et al., 2001) et les topoisomérases (Topcu et Castora, 1995) sont également susceptibles de jouer un rôle dans ce processus. Il existe également des preuves expérimentales suggérant que le noyau cellulaire peut exercer un contrôle négatif sur le génome mitochondrial par l’intermédiaire de certaines substances nucléaires à courte durée de vie (Rinaldi et al., 1979).
ADN Polymérase γ
La surexpression de la sous-unité catalytique (sous-unité A) de l’ADNm polymerase γ dans des cellules d’insecte ou humaines cultivées n’a pas altéré les taux d’ADNmt (Lefai et al., 2000a, Spelbrink et al., 2000). Cependant, pour les mouches transgéniques surexprimant pol γ -A, le nombre de génomes mitochondriaux a été réduit de façon drastique, indiquant que bien que les cellules puissent tolérer une quantité variable de la sous-unité catalytique pol γ dans certaines conditions, les niveaux de sous-unité A pourraient être critiques dans le contexte. de l’organisme entier (Lefai et al., 2000b). Les mouches avec des mutations dans pol γ -A montrent des problèmes avec le système visuel et un comportement altéré dans le stade errant, les deux semblant être une conséquence des défauts de locomotion (Iyengar et al., 1999). L’expression de la sous-unité accessoire de pol γ (sous-unité B) semble mieux corrélée avec l’activité de réplication de mtDNA. Le niveau d’équilibre de l’ARNm de pol γ -B augmente pendant les premières heures de développement, atteignant sa valeur maximale au début de la réplication de l’ADNmt chez les embryons de Drosophila. Ce profil d’expression n’a pas été observé avec l’ARNm de pol y -A (Lefai et al., 2000b). Un lien potentiel entre la réplication nucléaire et mitochondriale de l’ADN a également été décrit chez la drosophile. Le promoteur pol γ -B contient un site apparenté à la réplication de l’ADN (DRE), précédemment identifié dans les gènes impliqués dans la réplication de l’ADN nucléaire, ce qui suggère un mécanisme régulateur commun contrôlant la réplication nucléaire et mitochondriale de l’ADN (Lefai et al. , 2000b). Récemment, une mutation de la sous-unité A de la polynucléotide A a été associée à de multiples délétions de l’ADN mitochondrial chez des patients atteints d’ophtalmoplégie externe progressive (Goethem et al., 2001).
